BIOLOGÍA Y GEOLOGÍA















Para ir a donde no se sabe hay que ir por donde no se sabe.” San Juan de la Cruz
“It must be a strange world not being a scientist, going through life not knowing--or
maybe not caring about where the air came from, where the stars at night came from or
how far they are from us. I WANT TO KNOW” Michio Kaku
“Nullius in verba” Robert Boyle, Christopher Wren y Robert Hooke


GENÉTICA MOLECULAR
SÍNTESIS DE PROTEÍNAS
(TRADUCCIÓN)
1. TRADUCCIÓN DEL ARNm
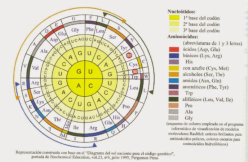
La traducción es el paso del mensaje genético del ARNm (codificado como nucleótidos) al de las proteínas (codificado como aminoácidos). Para poder pasar de un tipo de molécula a otro se precisa un código que relacione los nucleótidos con los aminoácidos. Ese código es el código genético.1.2. EL CÓDIGO GENÉTICO
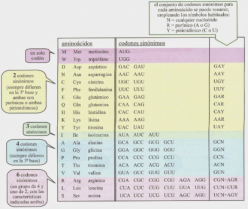
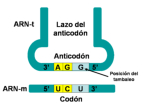
El código genético es una correspondencia entre nucleótidos y aminoácidos, que permite pasar el mensaje contenido en los genes hasta las proteínas. En el ARNm, los aminoácidos se codifican mediante “palabras” de tres letras, que son los tripletes de bases o codones. Sin embargo, no existe ninguna relación química entre los codones y los aminoácidos y, por tanto, los aa no pueden unirse directamente al ARNm. Por ello se necesitan “intérpretes” que conviertan un lenguaje en otro. Dichos intérpretes son las moléculas de ARNt. CARACTERÍSTICAS DEL CÓDIGO GENÉTICO Es universal: salvo raras excepciones (mitocondrias, ciliados), todos los seres vivos tienen el mismo, y no es ambiguo (cada codón significa siempre lo mismo). Es degenerado o redundante. Existen 4 3 =64 tripletes posibles y sólo 20 aminoácidos, por lo que cada aa puede estar codificado por más de un codón (codones sinónimos). Todos los codones tienen sentido y se leen en el ARNm en sentido 5’ 3'. Carece de solapamiento: los codones no comparten bases entre sí y se colocan uno a continuación de otro.2. PAPEL DE INTÉRPRETE DE LOS ARNt
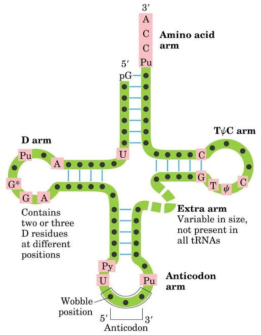
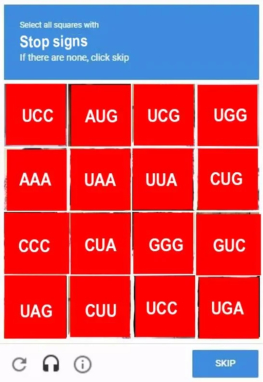
Los ARNt son los que mantienen la correspondencia entre los tripletes del ARNm y los aminoácidos de las proteínas. Esta correspondencia depende de dos tipos de adaptaciones o reconocimientos: Primera adaptación: cada ARNt debe unirse de forma específica a un determinado aa. Para ello precisa de la enzima aminoacil ARNt sintetasa, que presenta dos sitios activos específicos. Uno reconoce al aminoácido (existen al menos 20 enzimas diferentes) y el otro reconoce el bucle o brazo D del ARNt correspondiente. La enzima cataliza la unión del grupo carboxilo (-COOH) del aa con el hidroxilo (-OH) del brazo aceptor del ARNt (extremo CCA-OH). Segunda adaptación: se produce en los ribosomas. Los ARNt reconocen los codones del ARNm y se unen a ellos mediante su anticodón correspondiente. En este proceso los ribosomas actúan como adaptador. La unión codón-anticodón sólo es estricta para las dos primeras bases del anticodón del ARNt. La 3ª base, la del extremo 5’, puede unirse a diferentes bases. Esto se denomina balanceo y es la causa de la degeneración del código. Por ello, existen entre 35 y 50 ARNt diferentes. Se llaman ARNt isoaceptores a los ARNt que aceptan el mismo aa.3. TRADUCCIÓN EN EUCARIOTAS
La traducción es muy similar en procariotas y eucariotas y consta de 3 etapas: iniciación, elongación y terminación.3.1. INICIACIÓN
Consiste en la formación del complejo de iniciación 80S, formado por un ribosoma unido al ARNm y al ARNt i met (ARNt iniciador cargado con metionina). Para ello se requieren varias fases: a. Diversos factores de iniciación (FI) y la energía del GTP permiten la unión de la subunidad pequeña del ribosoma (40S) con el ARNt i met . b. El complejo formado reconoce la “caperuza” de metil guanosina trifosfato del ARNm gracias a otros FI, uniéndose al extremo 5’ del ARNm. c. Mediante energía en forma de ATP, la subunidad pequeña del ribosoma se desplaza por el ARNm en dirección 5’ 3' hasta que encuentra la primera secuencia AUG, que es el codón de iniciación (y que codifica para metionina). El ARNt i met se une entonces a dicho codón, ya que posee el anticodón correspondiente: UAC. d. Los FI se liberan y se acopla la subunidad mayor (60 S), con lo que se forma el definitivo complejo de iniciación 80 S. La subunidad mayor presenta tres hendiduras o sitios de fijación: el E (de liberación); el P (de fijación del ARNt unido a la cadena peptídica) y el A (de fijación de un segundo ARNt que trae un nuevo aa). La traducción comienza siempre, por tanto, por el codón AUG del ARNm más cercano a la “caperuza” y, por tanto, inicialmente todas las cadenas peptídicas comienzan por el aa metionina, aunque luego puede ser eliminado.3.2. ELONGACIÓN
La elongación es el proceso por el que se sintetiza la cadena peptídica dentro del ribosoma, catalizado por la enzima peptidil transferasa. El proceso consta de tres fases sucesivas que se repiten para cada uno de los aa añadidos a la cadena peptídica. Primera fase: entrada del ARNt-aminoácido: el sitio P está ocupado por el ARNt i met , mientras los sitios E y A están vacíos. Ahora el sitio A es ocupado por el ARNt cuyo anticodón es complementario del siguiente triplete en el ARNm. Esto se consigue con el factor de elongación FE-1 y energía del GTP. Segunda fase: formación del enlace peptídico. La peptidil transferasa rompe el enlace de la metionina unida al ARNt i met del sitio P y la une, mediante enlace peptídico, al que transporta el ARNt del sitio A. Esta labor está catalizada también con ayuda de ribozimas del propio ribosoma. De esta forma se obtiene un dipéptido unido al ARNt del sitio A. Tercera fase: traslocación. El factor de elongación FE-2, con energía del GTP, mueve el ribosoma a los largo del ARNm exactamente tres nucleótidos en dirección 5’ 3'. De esta forma: El sitio P queda ocupado por el ARNt con el dipéptido. El sitio A queda vacío y en espera de alojar un nuevo ARNt. El sitio E queda ocupado por el ARNt i ya sin aa. Repetición de las tres fases: la entrada de un nuevo ARNt aa en el sitio A expulsa al ARNt del sitio E y se repiten otra vez las tres fases. De esta manera, se forma un tripéptido, luego un tetrapéptido, etc.3.3. TERMINACIÓN
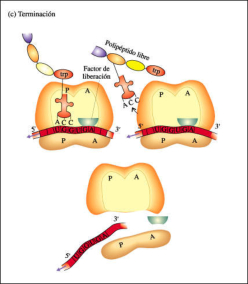
La terminación tiene lugar cuando tras una traslocación aparece en el sitio A uno de los tres codones de terminación: UAA, UAG o UGA. Un factor proteico de terminación (RF) se une a dicho codón e impide la unión de un aminoacil-ARNt en el sitio A. La enzima peptidil transferasa cataliza entonces la transferencia de la cadena peptídica a una molécula de agua, lo que provoca la hidrólisis entre la cadena peptídica y el ARNt que lo portaba. La cadena peptídica queda así liberada; las dos unidades del ribosoma se separan y el ARNm puede volver a traducirse o es degradado.3.4. MADURACIÓN POSTRADUCCIONAL
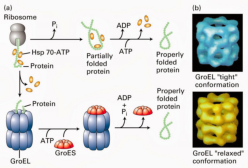
En general, los polipéptidos recién sintetizados no son funcionales: precisan de ciertas modificaciones que suponen un proceso de maduración postraduccional: Plegamiento de las proteínas: tras su síntesis, las cadenas peptídicas han de adquirir su estructura tridimensional para ser activas. Para ello se forman puentes disulfuro y otras uniones, así como la asociación con otras cadenas para adquirir estructura cuaternaria. En el proceso de plegamiento y tránsito de proteínas colaboran unas proteínas especiales llamadas chaperonas moleculares. Adición de grupos prostéticos: glúcidos (glucoproteínas), lípidos (lipoproteínas), grupos hemo, coenzimas, minerales, etc. Modificación de algunos aminoácidos: fosforilación, metilación, hidroxilación, etc. Cortes proteolíticos: eliminación de algunos aminoácidos de la cadena, como la metionina inicial o aminoácidos intermedios (como en la insulina).4. REGULACIÓN DE LA EXPRESIÓN GÉNICA
Los organismos han de regular su actividad metabólica en función de los estímulos que les llegan del medio. Esta regulación puede hacerse de dos formas básicas: modificando la actividad enzimática (cambios de pH y temperatura, alosterismo, compartimentación celular, ...) o modificando la concentración de enzimas (mediante la regulación de la expresión génica). La regulación de la expresión génica en eucariotas es mucho más compleja y menos conocida que en procariotas.4.1. REGULACIÓN GÉNICA Y DIFERENCIACIÓN CELULAR
Una razón de la supervivencia de los organismos es su capacidad para controlar la síntesis proteica. Sólo se sintetizan aquellas proteínas que se necesitan y en el momento necesario. Esta es igualmente la clave de la diferenciación celular: en cada tejido las células sólo expresan aquellas proteínas que precisarán para sus funciones, aunque contengan todo el ADN del organismo. Las células madre se diferencian por su capacidad para producir uno o más tipos de tejidos. Las células madre se encuentran en todos los organismos vivos pluricelulares. También son conocidas como células stem, por su denominación en inglés, o células troncales, ya que la traducción del término stem es tronco. Su característica principal es que al reproducirse mediante el proceso de mitosis (se dividen en dos células) pueden formar más células madre o diferenciarse, es decir, transformarse en células especializadas, de tal modo que son esenciales para la renovación de las células de diferentes tejidos y para la reparación de los mismos. Si se tiene en cuenta su potencial para generar diferentes tipos de células, se clasifican del modo siguiente: Totipotentes. En realidad son las células madre embrionarias, pues se forman en el momento en el que el óvulo se une con un espermatozoide para formar el cigoto y pueden generar cualquier tipo de célula, tejido u órgano del cuerpo. Pluripotentes o pluripotenciales inducidas (IPS). Son capaces de generar prácticamente cualquier tipo de células En realidad no son células madre adultas, sino que se obtienen a partir de células diana reprogramadas genéticamente. Son muy similares a las células madre embrionarias. Multipotentes. Se generan a partir de las pluripotentes y se pueden encontrar en los tejidos nuevos, lo que supone que ya han sufrido una primera diferenciación y que continúan haciéndolo. Son muy adecuadas para su uso en trasplantes de tejidos, ya que reducen significativamente las posibilidades de rechazo. Unipotentes. Puede generar un único tipo de células y surgen a partir de las células madre pluripotentes. Un ejemplo es el de las células de la piel, que están en continuo proceso de renovación, por lo que si se extrae un trozo de piel no dañada se pueden cultivar las células hasta generar tejido suficiente para poder trasplantarlo a un paciente que ha sufrido quemaduras importantes.4.2. REGULACIÓN DE LA EXPRESIÓN GÉNICA EN
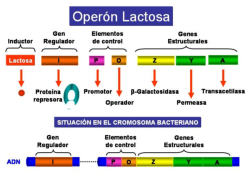
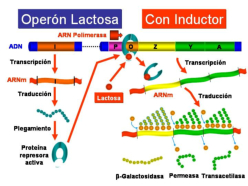
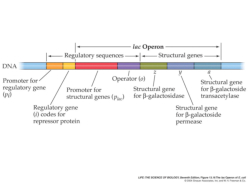
PROCARIOTAS: EL OPERÓN LAC
La regulación en procariotas tiene lugar, principalmente, en la transcripción. Para ello utilizan proteínas reguladoras codificadas por genes reguladores. Dichas proteínas pueden actuar como controles negativos, reprimiendo la transcripción (sistema reprimible); o como controles positivos, intensificándola (sistema inducible). Jacob, Monod y colaboradores analizaron el sistema de la lactosa en E. coli, de manera que los resultados de sus estudios permitieron establecer el modelo genético del operón que permite comprender cómo tiene lugar la regulación de la expresión génica en bacterias. Jacob y Monod recibieron en 1965 el Premio Nobel por estas investigaciones (The PaJaMo experiment). Un operón es grupo de genes estructurales cuya expresión está regulada por los mismos elementos de control (promotor y operador) y genes reguladores. Los principales elementos que constituyen un operón son los siguientes: Los genes estructurales: llevan información para polipéptidos. Se trata de los genes cuya expresión está regulada. Los operones bacterianos suelen contener varios genes estructurales, son poligénicos o policistrónicos. Hay algunos operones bacterianos que tienen un solo gen estructural. Los operones eucarióticos suelen contener un sólo gen estructural siendo monocistrónicos. El promotor (P): se trata de un elemento de control que es una región del ADN con una secuencia que es reconocida por la ARN polimerasa para comenzar la transcripción. Se encuentra inmediatamente antes de los genes estructurales. Abreviadamente se le designa por la letra P. El operador (O): se trata de otro elemento de control que es una región del ADN con una secuencia que es reconocida por la proteína reguladora. El operador se sitúa entre la región promotora y los genes estructurales. Abreviadamente se le designa por la letra O. El gen regulador (i): secuencia de ADN que codifica para la proteína reguladora que reconoce la secuencia de la región del operador. El gen regulador está cerca de los genes estructurales del operón pero no está inmediatamente al lado. Abreviadamente se le denomina gen i. Proteína reguladora: proteína codificada por el gen regulador. Está proteína se une a la región del operador. Inductor: sustrato o compuesto cuya presencia induce la expresión de los genes. El operón lactosa, que abreviadamente se denomina operón lac, es un sistema inducible que está bajo control negativo, de manera que la proteína reguladora, producto del gen regulador i, es un represor que impide la expresión de los genes estructurales en ausencia del inductor. El inductor del sistema es la lactosa. Los genes estructurales del operón lactosa (necesarios para degradar la lactosa) codifican para la β-galactosidasa, que cataliza la hidrólisis de la lactosa en glucosa más galactosa; la galactósido permeasa, que transporta galactósidos al interior de la célula bacteriana; y una acetiltransferasa, que impide que se metabolicen otros galactósidos que no sean la lactosa. En ausencia del inductor (la lactosa), la proteína represora, producto del gen i, se encuentra unida a la región operadora e impide la unión de la ARN-polimerasa a la región promotora y, como consecuencia, no se transcriben los genes estructurales. Sin embargo, en presencia del inductor (la lactosa), este se une a la proteína reguladora que cambia su conformación y se suelta de la región operadora dejando acceso libre a la ARN-polimerasa para que se una a la región promotora y se transcriban los genes estructurales. Por consiguiente, la presencia del inductor hace que se expresen los genes estructurales del operón, necesarios para metabolizar la lactosa.4.3. REGULACIÓN DE LA EXPRESIÓN GÉNICA EN
EUCARIOTAS
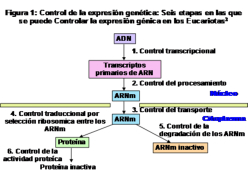
En eucariotas la regulación de la expresión génica es mucho más compleja y menos conocida. Básicamente tiene lugar en cinco niveles: 1. Estructura de la cromatina: se controla mediante metilación del ADN (silencia la expresión); metilación de histonas (condensa la cromatina e impide su transcripción) y acetilación de histonas (descondensa la cromatina y favorece la transcripción). 2. Control de la transcripción: mediante elementos traslocables o genes saltarines, como transposones y retrotransposones, que pueden inactivar o sobreactivar la transcripción; y mediante los factores de transcripción (potenciadores o enhancers e inhibidores o silencers). 3. Control de la maduración postranscripcional: splicing alternativo. 4. Control de la traducción: degradación de los ARNm, actuación de ARNi (ARN de interferencia), etc. 5. Control del proceso postraduccional: maduración, cambios conformacionales, degradación por el proteasoma, etc.































ARN captcha






































![Cerrar [x]](bg2bto_htm_files/close.png)