

BIOLOGÍA Y GEOLOGÍA
GENÉTICA MOLECULAR.
REPLICACIÓN DEL ADN
1.
BIOSÍNTESIS DE ADN
Durante la división celular las células hijas reciben una copia del material genético de la célula madre. Para que esto sea posible se requiere que el ADN se duplique previamente. El proceso de copia del ADN se denomina duplicación o replicación. El proceso es similar en procariotas y eucariotas. En la replicación del ADN las dos hebras se separan y cada una actúa de molde para la síntesis de una nueva cadena con una secuencia de bases complementaria de la original. La replicación del ADN sigue un modelo semiconservativo: en cada nueva doble hélice una cadena es la original y la otra es una réplica.2.
REPLICACIÓN DEL ADN EN BACTERIAS
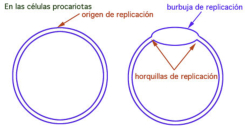
Las bacterias presentan un único cromosoma de ADN circular. La replicación comienza en un punto donde se produce una burbuja de replicación, que se extiende y forma dos horquillas de replicación en las que cada hebra sirve de molde para sintetizar la complementaria. Las horquillas se extienden en sentidos opuestos hasta que se encuentren, momento en que se separan los dos nuevos cromosomas. El proceso consta de 3 etapas: iniciación (apertura y desenrollamiento de la doble hélice), elongación (síntesis de dos nuevas cadenas de ADN) y corrección de errores.2.1. INICIACIÓN: APERTURA Y DESENROLLAMIENTO DE
LA DOBLE HÉLICE
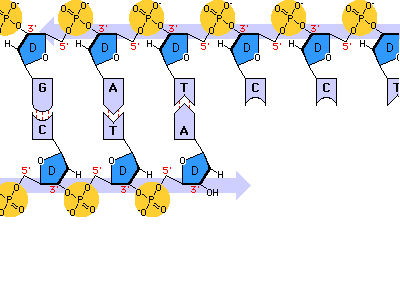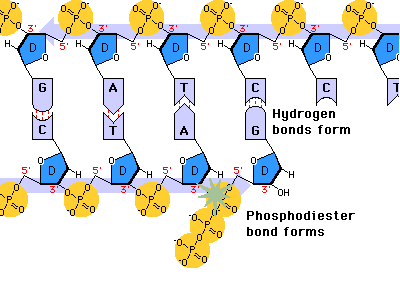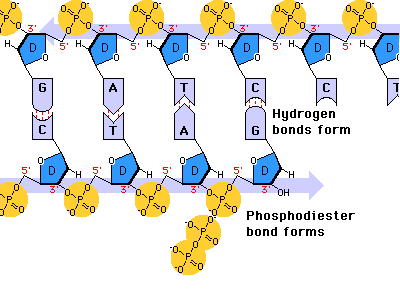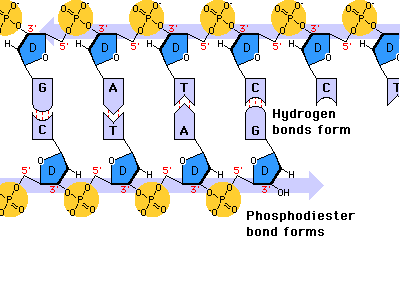
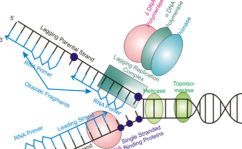
En la bacteria Escherichia coli la separación de las dos cadenas de ADN comienza en un punto concreto del cromosoma, rico en secuencias GATC, llamado oriC, que marca el origen de la replicación. A partir de este origen se forma una estructura con ADN abierto llamada burbuja de replicación, que se extiende en sentidos opuestos separando las dos hebras del ADN en forma de “Y”: son las horquillas de replicación. En las horquillas el ADN está abierto y cada hebra actúa como molde para la síntesis de ADN. Por ello se dice que la replicación es bidireccional. Para replicar el ADN, además de la ADN polimerasa, se precisan un conjunto de proteínas y enzimas que desenrollan las hebras: el replisoma. Primero intervienen las helicasas, que abren la doble hélice como una cremallera. Al mismo tiempo, para evitar tensiones por la torsión de las hebras, actúan girasas y topoisomerasas. Seguidamente, las proteínas SSBP (single-strand binding protein o proteínas de unión a cadena sencilla) se unen a cada hebra de ADN para estabilizarlo y evitar que se vuelvan a enrollar.2.2. ELONGACIÓN: SÍNTESIS DE DOS NUEVAS CADENAS
DE ADN
Una vez formada la burbuja de replicación intervienen las enzimas ADN polimerasas, encargadas de hacer copias de cada cadena de ADN. En E. coli existen 3 enzimas: las ADN polimerasas I y III, que replican y corrigen errores, y la ADN polimerasa II, que repara daños. La principal enzima es la ADN polimerasa III que: Recorre las hebras molde en dirección 3’→5’ y selecciona el desoxirribonucleótido trifosfato que debe ser complementario a la base del molde. Si el nucleótido trifosfato seleccionado es, efectivamente, el complementario, cataliza su hidrólisis, separa el pirofosfato (PP) del nucleótido y éste es unido a la cadena de ADN mediante enlace fosfodiéster gracias a la energía de la hidrólisis. PROBLEMAS QUE DEBE RESOLVER LA ADN POLIMERASA III En primer lugar, la ADN polimerasa III es incapaz de iniciar la síntesis de ADN por sí misma, sólo pueden alargar una cadena ya empezada y que tenga un extremo 3’ (OH) libre. Debido a ello, para comenzar la síntesis actúa primero una enzima ARN polimerasa (primasa), que coloca al comienzo de la cadena un pequeño fragmento de ARN llamado cebador (primer) que aporta el extremo hidroxilo 3’ libre. El cebador será después eliminado. En segundo lugar, la ADN polimerasa sólo puede leer las hebras molde en sentido 3’→5’ al tiempo que sintetiza la nueva hebra en sentido 5’→3’. La cadena molde 3’→5’ se lee y se replica de manera continua, por lo que se llama hebra conductora o líder. Sin embargo, la cadena antiparalela no puede leerse ni replicarse de forma continua. Para replicarla, la doble hélice se abre lo suficiente como para que la ADN polimerasa III pueda sintetizar pequeños fragmentos de ADN en dirección 5’→3’ (fragmentos de Okazaki), que luego se unirán para formar la hebra retardada, llamada así porque tarda más en sintetizarse. PROCESO DE SÍNTESIS DE ADN En conjunto, la replicación del ADN puede resumirse así: Hebra conductora o líder: se sintetiza de forma continua. En la horquilla de replicación, la ARN polimerasa (primasa) sintetiza un pequeño fragmento de ARN (cebador o prímer). Este fragmento es alargado de forma continua por la ADN polimerasa III. Posteriormente, la ADN polimerasa I eliminará el cebador y lo sustituirá por ADN. Finalmente, una ADN ligasa unirá ambos fragmentos. Hebra retardada: se sintetiza de forma discontinua, en fragmentos llamados fragmentos de Okazaki . Cada fragmento se forma así: La ARN polimerasa (primasa) fabrica un fragmento de ARN cebador (prímer). El cebador es alargado por la ADN polimerasa III que fabrica un pequeño fragmento de ADN: fragmento de Okazaki. La ADN polimerasa I actúa como exonucleasa y elimina el cebador. La propia polimerasa I rellena el hueco con ADN complementario al molde. Finalmente, la ADN ligasa une los diferentes fragmentos para dar la hebra retardada.2.3 CORRECCIÓN DE ERRORES
La actividad de las polimerasas debe ser rápida y fiel, de forma que la información genética pase con fidelidad de generación en generación. Por ello, en el replisoma, el conjunto de más 50 proteínas que intervienen en la replicación, cuenta con numerosas enzimas correctoras. Estas enzimas detectan y corrigen las secuencias erróneas. Existen 2 tipos de mecanismos correctores: corrección de pruebas y corrección postreplicativa. CORRECCIÓN DE PRUEBAS: ACTIVIDAD AUTOCORRECTORA DE LA ADN POLIMERASA La actividad de las ADN polimerasas I y III en E. coli conlleva un error de apareamiento cada 10 6 nucleótidos. Sin embargo, ambas enzimas, además de polimerasa tienen actividad exonucleasa. Así, después de cada proceso de polimerización, estas enzimas “comprueban” el nucleótido adicionado y, si es incorrecto, lo eliminan y sustituyen por el adecuado. El uso de cebadores parece ser que contribuye también a cometer menos errores. La actividad correctora reduce las probabilidades de error a uno por cada 10 8 pares de bases. CORRECCIÓN POSTREPLICATIVA Además de la corrección de pruebas existe un mecanismo corrector postreplicativo. Este mecanismo lo realizan enzimas correctoras del replisoma. En primer lugar, debe reconocerse la cadena de nueva síntesis, donde está el error, y distinguirla de la cadena molde original. Esto se consigue porque en la cadena molde, las secuencias GATC tienen la A metilada, mientras que en la réplica aún se tarda un tiempo en metilarse. Luego se elimina el error cortando un fragmento del ADN que lo contiene mediante una endonucleasa. Finalmente, la ADN polimerasa I rellena el hueco y una ligasa unirá los fragmentos. La corrección postreplicativa reduce la probabilidad de error a uno por cada 10 10 pares de bases.3. REPLICACIÓN DEL ADN EN EUCARIOTAS
La replicación en eucariotas se produce en la fase S del ciclo celular y es similar a la de procariotas, pero mucho más compleja, por lo que presenta similitudes y diferencias: A. SIMILITUDES Es semiconservativa y bidireccional. Existe una hebra conductora, que se sintetiza de forma continua, y una hebra retardada, que se sintetiza de forma discontinua mediante fragmentos de Okazaki. Se precisa de cebadores para iniciar la síntesis de las cadenas. B. DIFERENCIAS La replicación en eucariotas es mucho más lenta: unas decenas de nucleótidos por segundo frente a los 1.000 de E. coli. Sólo se realiza en la fase S de la interfase celular. En eucariotas hay 5 ADN polimerasas: α (sintetiza el cebador y la hebra retardada); β (ligasa: une fragmentos de Okazaki); γ (replica el ADN mitocondrial); δ (sintetiza la hebra conductora); ε (polimeriza los fragmentos de Okazaki). Los fragmentos de Okazaki son más pequeños. El ADN de eucariotas está asociado a histonas, que también han de duplicarse. Las histonas antiguas permanecen en la hebra conductora y se crean otras nuevas para la retardada. El genoma eucariota es mucho mayor que el de los procariotas. Consta de numerosas cadenas lineales de ADN. Dado que, además, la replicación es más lenta, duplicar todo el ADN con el modelo procariota tardaría semanas. Para hacerlo en sólo unas horas, los eucariotas presentan numerosas burbujas de replicación. En humanos puede haber unos 30.000 puntos de inicio.3.1. LOS TELÓMEROS: ENVEJECIMIENTO Y APOPTOSIS
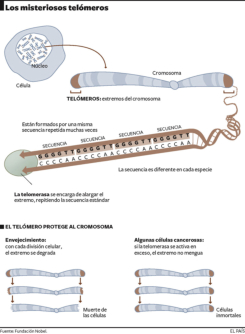
Los cromosomas eucariotas son lineales y en sus extremos tienen secuencias repetitivas de bases TTAGGG llamadas telómeros (en humanos y murinos hasta 2.000 repeticiones) Los telómeros protegen los extremos de los cromosomas, impidiendo que se unan a otras cadenas de ADN y evitando la pérdida de información importante debido a un problema en los extremos 5’ de los cromosomas. Este problema consiste en que, al eliminar el cebador del extremo 5’ de las hebras sintetizadas, el hueco no se puede rellenar, pues carece de extremo OH 3’ sobre el que actúen las polimerasas. Como consecuencia, en cada ciclo de replicación se pierde parte del telómero. Tras un número determinado de divisiones, los telómeros han desaparecido y quedan al descubierto los extremos de los cromosomas. Para evitar la pérdida de información, la célula activa el mecanismo de apoptosis que provoca la muerte celular. Por tanto, las células sólo pueden dividirse un número determinado de veces antes de morir, lo que está relacionado con el envejecimiento. Los telómeros actúan como un “reloj molecular”. Sin embargo, en ciertas células (células madre, células cancerígenas) los telómeros pueden durar indefinidamente gracias a una enzima llamada telomerasa, que convierte a estas células en virtualmente inmortales. La telomerasa es una ribonucleoproteína que actúa como retrotranscriptasa o transcriptasa inversa, tiene una secuencia de ARN molde del ADN telomérico. Así, es capaz de alargar los telómeros tras cada división celular.







































Ciencia al día
En junio de 2016 se publicaba un artículo sobre el descubrimiento de un nuevo elemento origen en
bacterias. Se trata del AdnA-trio, un origen de replicación que se halla en las hebras simples de ADN
una vez abierta la hélice. Consiste en un motivo trinucleótido repetido donde se unen las AdnA para su
estabilización como hebra sencilla. (http://www.nature.com/nature/journal/vaop/ncurrent/full/nature17962.html)

Para ir a donde no se sabe hay que ir por donde no se sabe.” San Juan de la Cruz
“It must be a strange world not being a scientist, going through life not knowing--or
maybe not caring about where the air came from, where the stars at night came from or
how far they are from us. I WANT TO KNOW” Michio Kaku
“Nullius in verba” Robert Boyle, Christopher Wren y Robert Hooke



Dibujo de la la telomerasa humana realizado por Eva Nogales. Su estructura
general es bilobal (con dos lóbulos) y consiste en un ARN extendido que
actúa como andamio para varias proteínas. El lóbulo H/ACA facilita el
ensamblaje de la telomerasa a partir de sus componentes y controla el tráfico
en el núcleo. El otro lóbulo contiene el núcleo catalítico de la enzima.

EVA NOGALES
La española que ha logrado retratar a la enzima de la inmortalidad
Vídeo sobre la telomerasa (en inglés)
Apoptosis de una neurona
















































![Cerrar [x]](bg2bto_htm_files/close.png)




